Universidad de Valencia
Facultad de Medicina & Odontología
Diseño y presentación de trabajos científicos

Sofía Milla Herranz
Valencia 28/02/2008
ÍNDICE
Portada
Resumen
Palabras clave
Introducción
El código genético
-
*Degenerado
- *Universal
- *No se solapa
- La maquinaria de la síntesis de proteínas
- *ARNt
- *Formación de aminoacil-tRNA
- * Síntesis de las proteínas en los ribosomas
- * El proceso de la traducción:
- -Inicio
- -Elongación
- -Terminalización
Bibliografía
Bibliografía complementaria
Glosario
Apéndice biográfico
RESUMEN
La síntesis de proteínas constituye la culminación de la transferencia de información
genética desde el ADN a las proteínas. La información deberá pasar desde el lenguaje
del ADN al del RNA y este al lenguaje de los 20 aminoácidos de las proteínas.
El código genético, especifica un aminoácido para cada codón. El RNAt es el puente que
une los dos útimos lenguajes gracias al anticodón que interactúa con los codones
específicos del RNAm, y con los aminoácidos gracias al locus de unión en el extremo 3´
de la molécula. El inicio implica el ensamblaje del ribosoma y el RNAt cargado con el
codón de iniciación del RNAm, gracias a factores de iniciación y GTP. El ensamblaje se
trata de uniones peptídicas entre aminoácidos que formarán el polipéptido con la ayuda
también en este caso de factores de elongación y GTP. La finalización ocurre cuando el
ribosoma llega a un codón de interrupción y los factores de liberación catalizan la
liberación de una proteína. Después de eso, la proteína tomará su debida conformación
y se hidrolizarán los metabolitos que han tomado parte en el proceso.
PALABRAS CLAVE
Locus A, locus P, RNAt, mRNA, codón.
INTRODUCCIÓN
Las proteínas, por su tamaño, no pueden atravesar la membrana plasmática de la célula
por eso es que existe en su interior un mecanismo que las construye (síntesis) según las
necesidades que tenga en ese momento la célula. (Sokolovsky S, 2005)
La síntesis de proteínas consta en realidad de dos etapas: la primera etapa (transcripción)
ocurre dentro del núcleo de las células eucariotas, aquí la secuencia de nucleótidos que
denominamos gen (segmento de ADN que determina una proteína) se transcribe en unamolécula de ARN. Posteriormente, en la segunda etapa (traducción - síntesis de proteína
propiamente dicha) el ARN pasa del núcleo al citoplasma donde es traducida por los ribosomas que sintetizan una proteína.
Este proceso es de fundamental importancia ya que básicamente todos los caracteres
que la célula presenta, está regulado por la suma de sus actividades enzimáticas.
En pocas palabras, todo lo que la célula es y puede realizar depende de la acción enzimática
específica. Por eso, es importante tener algunos conceptos claros antes de entrar de lleno
al mecanismo de traducción de las proteínas.
CÓDIGO GENÉTICO
Cuando se piensa en la transferencia de la información de un RNA que contiene tan
sólo 4 bases diferentes (adenina, A; citosina, C; guanina, G; y uracilo, U) a una proteína
con 20 aminoácidos diferentes, resulta evidente que no existe una correspondencia
unívoca entre la secuencia de nucleótidos y aminoácidos. La secuencia del ARN se lee
de forma unidireccional por el ribosoma en “palabras” de letras (codones o tripletes)
formadas por tres bases consecutivas.(Lozano J & as, 2005, 361) Ésa es la forma para
especificar un aminoácido. Por lo tanto, al considerar todas las combinaciones de cuatro
nucleótidos tomados de tres en tres se tiene un total de 64 posibles codones. Tres de
estos codones (UAA, UAG, UGA) se utilizan como señales para interrumpir la síntesis
de una proteína. De este modo quedan 61 codones para especificar 20 aminoácidos.
>br>
- A. EL CÓDIGO ES DEGENERADO, AUNQUE INEQUÍVOCO
Una característica del código (conocida como degeneración) hace referencia al hecho
de que la mayor parte de los aminoácidos está codificada por más de un codón,
conociéndose como codones sinónimos el conjunto de codones diferentes que codifican
un mismo aminoácido. (Lozano J & as, 2005, 361) La inspección de una tabla de
codones (fig.1) demuestra que en la mayoría de los casos de varios codones para un
mismo aminoácido, la variación se produce en la tercera base del codón. (Smith C; Marks A; Lieberman M, 2006, 213)
- B. EL CÓDIGO ES CASI UNIVERSAL
A la vez, el código genético es universal, es decir, funciona por igual en todos los
sistemas biológicos, de modo que es el mismo para los virus, las bacterias, las células
eucarióticas. Sin embargo, se han constatado la existencia de algunas excepciones,
referentes al uso de unos pocos codones, en mitocondrias, cloroplastos y ciertos
protozoos.(Lozano J & as, 2005, 364)
- C. EL CÓDIGO NO ES SOLAPANTE Y NO TIENE PUNTUACIÓN
Hay que añadir que el mRNA no contiene puntuación para separar un codón del
siguiente y los codones no se solapan. Cada nucleótido se lee sólo una vez. (Smith C;
Marks A; Lieberman M, 2006, 214) Comenzando con un codón de iniciación (AUG)
cerca del extremo 5´ del mRNA, los codones se leen secuencialmente, finalizando con un codón de parada. (UGA, UAG o UAA) cerca del extremo 3´del mRNA.
* fig.1 Cada codón se refiere a un aminoácido. Viendo la gráfica se observa que el código genético es degenerado. (Devlin T, 2000, 722).
LA MAQUINARIA DE LA SÍNTESIS DE PROTEÍNAS
Los ribosomas están formados por una subunidad grande y otra pequeña que, al asociarse una con otra, presenta loci específicos en los que se fijan los RNAt. Estos loci se denominan aminoacilo o A y peptidilo o P. El locus A es aquel en que se sitúa una molécula de RNAt, que lleva el aminoácido apropiado en el tallo aceptor, antes de que éste se incorpore en la proteína. El locus P es la localización del ribosoma que contiene una molécula de RNAt con el polipéptido aminoterminal * de la proteína recién sintetizada unido aún a su tallo aceptor.( Baynes J; Dominiczak M, 2006, 457) Es precisamente en estos loci donde ocurre el proceso de formación del enlace peptídico. Este proceso está catalizado por la peptidil-transferasa, una enzima que forma el enlace peptídico entre el grupo amino del aminoácido en el locus A * y el extremo carboxilo terminal del péptido emergente unido al RNAt en el locus P*.
Existen 31 tipos diferentes de ARNt. Las moléculas intermediarias entre los codones del ARNm y los aminoácidos son los ARNt, los cuales tienen un dominio que se liga específicamente a uno de los 20 aminoácidos y otro que lo hace, específicamente también, con el codón apropiado. El segundo dominio consta de una combinación de tres nucleótidos -llamada anticodón* que es complementaria de la del codón.
Cada tipo de ARNt lleva antepuesto el nombre del aminoácido que transporta. (Sokolovsky S, 2005)por ejemplo, leucinil-ARNt para el aminoacil-ARNt de la leucina, lisinil-ARNt para el de la lisina, fenilalanil-ARNt para el de la fenilalanina, metionil-ARNt para el de la metionina, etcétera.
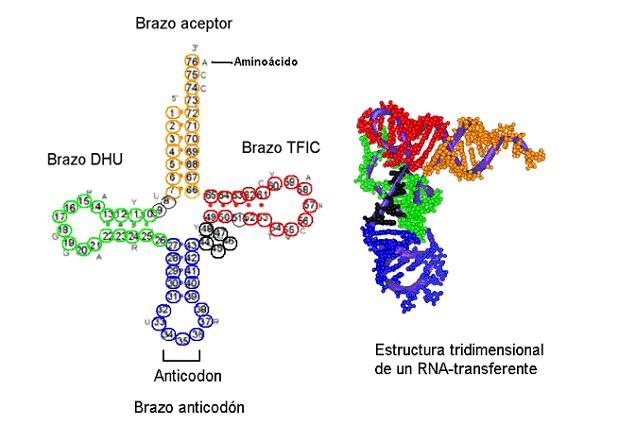
fig.2.Estructura molecular y tridimensional de la molécula de RNAt. (Devlin T, 2000, 725).
- A. FORMACIÓN DE AMINOACIL-tRNA
Existe también una molécula de RNAt distinta para cada uno de los codones. ( Baynes J; Dominiczak M, 2006, 457) El aminoácido es unido al tallo aceptor del RNAt por una enzima denominada aminoacil-RNAt-sintetasa; esta enzima cataliza la formación de un enlace éster entre el grupo 3´hidroxilo del nucleótido de adenosina del RNAt y el grupo carboxilo del aminoácido. Hay 20 sintetasas diferentes,una para cada aminoácido. (Smith C; Marks A; Lieberman M, 2006, 215) Cada sintetasa reconoce a un aminoácido específico y a todos los tRNA que transportan ese aminoácido.
La unión de un aminoácido a un RNAt es una reacción que consta de dos pasos. En primer lugar el grupo carboxilo del aminoácido es activado por reacción con trifosfato de adenosina (ATP) para formar un amino-aciladenilato intermedio que se fija al complejo de la sintetasa. En esta fase está listo para fijarse al locus A del ribosoma, donde con su aminoácido contribuye al crecimiento de la cadena peptídico.
Hay que citar que los aminoacil-ARNt sintetasas poseen capacidad autocorrectora de lectura de pruebas, rompiendo el enlace formado si la relación aminoácido ARNt no es correcta. (Lozano J & as, 2005, 365)
- B. SÍNTESIS DE LAS PROTEÍNAS EN LOS RIBOSOMAS
Los ribosomas desempeñan un papel fundamental en el proceso de unión de los aminoácidos para formar la cadena polipeptídica. Estás partículas están formadas por la interacción compleja de diferentes cadenas de ARNr y proteínas ribosomales, cuya composición más detallada se especifica en la figura 3.
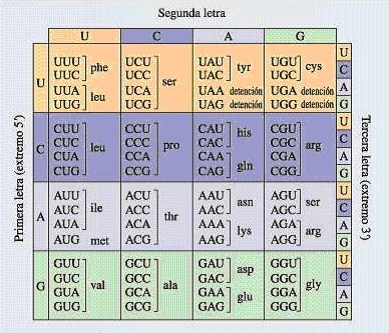
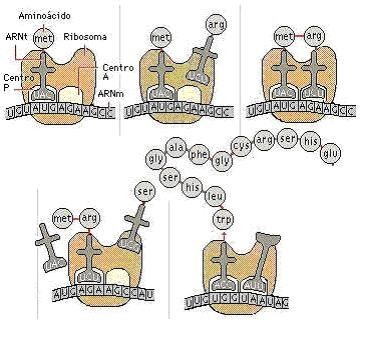
fig.3. El proceso de traducción se viene dado en los ribosomas, comenzando con
un codón de inicio y acabando con un codón de finalización. (Baynes J; Dominiczak M, 2006, 459).
Los ribosomas están formados por dos subunidades funcionales de tamaño diferente
(L, o mayor y S, o menor) que se asocian o se disocian a lo largo del proceso biosintético.
(Lozano J & as, 2005, 367) Aparte de los ribosomas, también participa en el proceso
una serie de factores proteicos de tranducción que interaccionan con moléculas de
aminoacil-ARNt, con los ribosomas y con el ARNm para llevar a cabo con precisión el
proceso de traducción.
En los ARNm de procariotas existe en la región 5´UTR y precediendo unos nucleótidos
al triplete de iniciación, la secuencia AGGAGGU, denominada secuencia de Shine Dalgarno*
que sirve como sitio inicial de anclaje del ARNm al ribosoma, a través de su
interacción con parte del extremo 3´de la molécula de ARNr 16S.(Lozano J & as, 2005, 367).
En el caso de las células eucariotas, el ribosoma se fija primero a la estructura de
7- metilguanina localizada en el extremo 5´del RNAm, y a continuación se desplaza por
la molécula hasta encontrar el primer codón AUG. E.( Baynes J; Dominiczak M, 2006, 459)
Ésto indica que al ribosoma ha de empezar la traducción por un residuo de metionina,
en el caso bacateriano siendo N-formil metionina.
- C. EL PROCESO DE LA TRADUCCIÓN
La traducción de una proteína comprende tres pasos: inicio, elongación y terminación.
Inicio: El inicio requiere la asociación de la subunidad pequeña del ribosoma (40S), del aminoácido N-terminal en forma de complejo aminoacil-tRNA, y el mRNA, todos en la orientación adecuada. A esta etapa sigue la asociación de la subunidad mayor (60S) para formar el complejo de incio completo sobre un ribosoma 80S. ( Devlin T, 2000, 725).El ribosoma ahora está completo. Contiene una subunidad grande y una pequeña y tiene lugares de unión para el tRNA, conocidos como los lugares P(peptidil) y A (aminoacil). Smith C; Marks A; Lieberman M, 2006, 216)
Este mecanismo está promovido por un grupo complejo de proteínas denominadas factotes de iniciación que participan sólo en la etapa de inicio. En las células procariotas participan 3 factores de iniciación, y en las eucariotas en cambio 12. La mayoría de los factores de iniciación ayuda a facilitar la asociación de la subunidad ribosómica pequeña con el RNAm y una molécula met-RNAt cargada. (Sokolovsky S, 2005)
Elongación: una vez completada la fase de iniciación, comienza el proceso de traducción de la información del RNAm a una proteína funcional. La elongación es un proceso secuencial de formación de enlaces peptídicos.( Devlin T, 2000 727). En cada paso la peptidil transferasa ribosómica transfiere el péptido creciente desde su tRNA transportador hasta el grupo alfa-amino del aminoácido que forma parte del aminoacil-tRNA especificado por el siguiente codón. En las células eucariotas, la RNAt cargado llega al ribosoma gracias a las acción de un factor de elongación denominado EF-1alfa.( Baynes J; Dominiczak M, 2006, 460). Para que sea activo el factor de elongación debe tener asociada una molécula de GTP. Cuando un complejo aminoacil-tRNA-EF1alfa_GTP se une a lugar A , el GTP se hidroliza a GDP. Esto provoca la disociación de EF1alfa-GDP del complejo ribosómico aminoacil-tRNA, permitiendo que continúe la síntesis de proteínas.(Smith C; Marks A; Lieberman M, 2006, 218)
En cambio, en procariotas el factor correspondiente de EF1alfa se llama EF-Tu.
Para formar el enlace peptídico en la primera ronda de elongación el aminoácido del tRNA en el lugar A forma un enlace peptídico con la metionina del tRNA en el lugar P. En las rondas siguientes, el aminoácido del tRNA en el lugar A forma un enlace peptídico con el péptido del tRNA en el lugar P. La peptidiltransferasa que no es una proteína sino el rRNA de la subunidad ribosómica grande, catalizará la formación del enlace actuando como ribozima. (Lozano J & as, 2005, 367)
El ciclo de elongación se repitede forma continua, deslizándose gradualmente el ribosoma sobre el ARNm en el sentido 5´->3´, hasta llegar a encontrar un triplete de terminación. Ello permite la interacción del complejo con algunos de los factores proteicos de terminación (FI) que se unen al sitio A gracias a su parecido estructural con los ARNt. Facilitando la liberación de la cadena polipeptídica sintetizada y la disociación del complejo biosintético tras la actuación de FI-3.
Durante todo el proceso, los tRNA que han ido viniendo, se aparearán con las bases del mRNA localizado en la subunidad menor del ribosoma.
Terminación: La presencia de un codón de terminación UAG, UAA o UGA en el sitio A del ribosoma no promueve la unión de ningún otro tRNA. En su lugar, otra proteína no ribosómica compleja, el factor de liberación (eRF) interacciona con el ribosoma, en forma de complejo eRF-GTP haciendo que en el locus P se libere la proteína unida a la última molécula de RNAt. La peptidil transferasa, actuando como hidrolasa, rompe el enlace éster entre el péptido y el tRNA y el polipéptido acabado es expulsado de su tRNA transportador y del ribosoma. ( Devlin T, 2000, 733)Después de liberarse de la proteína recién sintetizada, las subunidades ribosómicas, el RNAt y el RNAm se separan uno de otro. A continuación un factor de iniciación se une a la subunidad ribosómica pequeña y prepara la traducción de otro RNAm.
Englobando las 3 fases expuestas, queda citar que la traducción tiene un coste energético significativo. ( Devlin T, 2000 727) Para sintetizar un polipéptido se usa una cantidad considerable de energía. La activación del aminoácido convierte un ATP en AMP y pirofosfato, que se hidroliza normalmente en Pi. Otros dos enlaces de alta energía se hidrolizan por la acción de EF-1alfa y EF-2. Las modificaciones postraducción pueden añadirse al coste energético y por supuesto, también habría que sumar la energía necesaria para la biosíntesis de mRNA, tRNA, ribosomas y factores proteicos, aunque estos costes están distribuidos entre todas las proteínas formadas durante su periodo de existencia.( Devlin T, 2000 727)
Una vez liberado el polipéptido se plegará y adoptará una estructura tridimensional, definida en gran parte por la secuencia de aminoácidos. El pliegue vendrá a ser realizado gracias a unas proteínas auxiliares denominadas chaperonas (si el proceso no es satisfactorio se ubiquitizan y se destruyen en los llamados proteosomas* ). Antes de ser biológicamente activas, numerosas proteínas recién sintetizadas han de ser asimismo modificadas por diversos cambios químicos y estructurales, entrado en un proceso de post-traducción donde aquí no nos sumergiremos en él. Sólo citar que el aparato de Golgi tiene una importancia irrelevante sobre el tema.
En el proceso que acabamos de describir, el ribosoma se desplazaba a lo largo de una hebra de m-RNA leyendo los tripletes de uno en uno. La síntesis de proteínas progresa a razón de 15 aminoácidos/segundo.(Medciclopedia express, 2004)
Dada la longitud del m-RNA, varios ribosomas pueden ir leyendo codones y sintetizando proteínas. El conjunto se denomina poliribosoma.
A partir del anterior proceso se puede definir como gen un conjunto de nucleótidos de una molécula de DNA que sirve como molde para la producción de una proteína o una familia de proteínas si se producen operaciones de corte y empalme en el RNA.
BIBLIOGRAFÍA
Baynes J; Dominiczak M. 2006. BIOQUÍMICA MÉDICA 2º edición. España: Editorial El Sevier.
Devlin T. 2000. BIOQUÍMICA: LIBRO DE TEXTO CON APLICACIONES CLÍNICAS 3ª edición. Barcelona: Editorial Reverté S.A
Lozano J; Galindo J; García-Borrón J; Martínez J; Peñafiel R; Solano F. 2005. BIOQUÍMICA Y BIOLOGíA MOLECULAR PARA CIENCIAS DE LA SALUD 3º edición. Barcelona: Mc Graw Hill Interamericana.
Mediclopedia express
2004
SÍNTESIS DE PROTEÍNAS Y ÁCIDOS NUCLEICOS
Curso de fisiología
http://www.iqb.es/cbasicas/fisio/cap04/cap4_2.htm
consultado en: 17-4-2008
Smith C; Marks A; Lieberman M. 2006. BIOQUÍMICA BÁSICA de Marks, UN ENFOQUE CLÍNICO. Barcelona: Mc Graw Hill Interamericana.
Sokolovsky S
2005
Síntesis de Proteínas
Apuntes de silvia: matemáticas, física, química, biología
http://soko.com.ar/Biologia/sintesis_Proteinas.htm
consultado en : 17-4-2008
BIBLIOGRAFÍA COMPLEMENTARIA
Giasson BI, Lee VM-Y. Are Ubi1uination pathways central to Parkinson´s disease? Cell 2003; 114: 1-8
Sobre el ribosoma:
hhtp://www.resb.org/pdb/molecules/pdb10_1.htlm
Siegel V.A second signal recognition event required for translocation into de endoplasmic reticulum. Cell 1995; 82: 167-170
Sobre la ubiquitina:
http://www.notthingham.ac.uk/biochemcourses/students/ub/ubindex.htlm
GLOSARIO
Anticodón: secuencia de 3 bases en el tRNA , complementarias al codón del rna mensajero que determinarán un aminoácido.
Aminoterminal: extremo proteíco.
Codón: secuencia de 3 bases en mRNA que determinan un aminoácido.
Locus A: sitio del ribosoma que se asocia con el último aminoacil-trna que llega.
Locus P: sitio del ribosoma que se asocia con el peptidil-trna.
Proteosoma: organela especializada en digerir las proteínas en mal estado.
Shine Dalgarno: secuencia AGGAGGU, que sirve como sitio inicial de anclaje del ARNm al ribosoma.
APÉNDICE BIOGRÁFICO
Robert Kock: nació el 11 de diciembre de 1843 y murió el 27 de mayo de 1910. Fue un médico alemán. Se hizo famoso por descubrir el bacilo de la tuberculosis en (1882) así como también el bacilo del cólera en (1883) y por el desarrollo de los postulados de Koch.
De profesión médico, su primer descubrimiento fue el del bacilo de ántrax (agente del carbunco). En 1880 fue nombrado miembro del Departamento de Salud Imperial de Berlín, realizando aquí su descubrimiento más genial, el del bacilo de la tuberculosis (Bacilo de Koch). Probablemente tan importante como su trabajo en la tuberculosis, sean los llamados Postulados de Koch, que hace referencia al funcionamiento de la maquinaria de la síntesis de proteínas y cómo estas son las van a tener un principal protagonismo como principal antígeno en diferentes enfermedades.
La publicación más importante es la de los estudios sobre la tuberculosis, con la que recibió en premio Nobel en 1905.




